 A medida que aumenta la cantidad de datos genómicos, se hace cada vez más necesario disponer de métodos sistemáticos para organizarlos, explorarlos, compararlos, analizarlos y compartirlos. A pesar de ello, faltan plataformas adecuadas para satisfacer esta demanda.
A medida que aumenta la cantidad de datos genómicos, se hace cada vez más necesario disponer de métodos sistemáticos para organizarlos, explorarlos, compararlos, analizarlos y compartirlos. A pesar de ello, faltan plataformas adecuadas para satisfacer esta demanda.
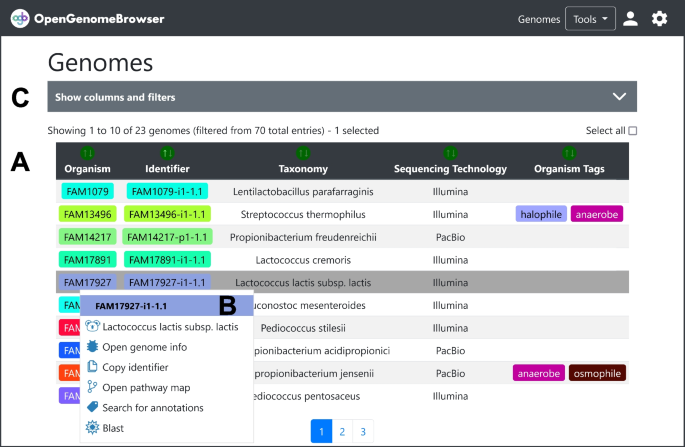
OpenGenomeBrowser es una plataforma autoalojable y de código abierto para gestionar el acceso a datos genómicos y simplificar drásticamente los análisis de genómica comparativa. Permite a los usuarios generar interactivamente árboles filogenéticos, comparar loci génicos, explorar rutas bioquímicas, realizar emparejamientos de rasgos génicos, crear diagramas de puntos, ejecutar búsquedas BLAST y acceder a los datos. Cuenta con un sistema flexible de gestión de usuarios, y su estructura modular de carpetas permite organizar los datos y metadatos genómicos y automatizar los análisis.
Los autores de este estudio explican que han probado OpenGenomeBrowser con genomas de bacterias, arqueas y levaduras. Proporcionan un contenedor de docker para simplificar la instalación y el alojamiento. El código fuente, documentación, tutoriales para OpenGenomeBrowser están disponibles en opengenomebrowser.github.io y un servidor de demostración es de libre acceso en opengenomebrowser.bioinformatics.unibe.ch.
Vea el artículo completo:



