 El análisis del compartimento progenitor hematopoyético humano se está transformando mediante enfoques multimodales unicelulares.
El análisis del compartimento progenitor hematopoyético humano se está transformando mediante enfoques multimodales unicelulares.
La indexación celular de transcriptomas y epítopos mediante secuenciación (CITE-seq) permite la elaboración de perfiles de proteínas de superficie acopladas y transcriptomas, revelando así los programas genómicos subyacentes a los estados progenitores.
Para realizar CITE-seq sistemáticamente en células primarias de la médula ósea humana, se utilizaron valoraciones con 266 anticuerpos CITE-seq (etiquetas derivadas de anticuerpos) y aprendizaje automático para optimizar un panel de 132 anticuerpos.
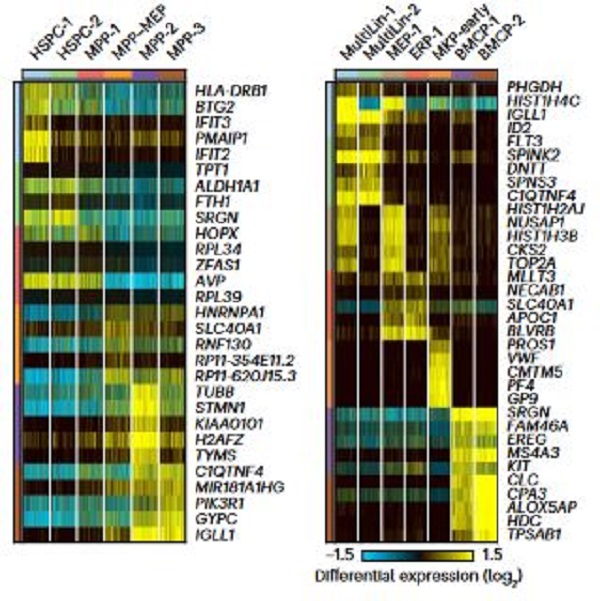
El análisis multimodal resolvió > 80 células madre, progenitoras, inmunes, estromales y de transición definidas por marcadores de superficie y transcriptomas distintivos. Este conjunto de datos permite soluciones de citometría de flujo para estados celulares predichos in silico e identifica docenas de marcadores de superficie celular detectados consistentemente en donantes que abarcan raza y sexo.
Finalmente, alineando las anotaciones de este atlas, se nominaron equivalentes de médula normal para poblaciones de células madre de leucemia mieloide aguda que difieren en la respuesta clínica. Este atlas sirve como recurso digital avanzado para el análisis de progenitores hematopoyéticos en la salud y las enfermedades humanas.
La combinación del análisis transcriptómico con la identificación de epítopes por secuenciación y técnicas de inteligencia artificial, permitió conformar un atlas de estados celulares y expresión de marcadores de superficies en progenitores hematopoyéticos humanos.
Los resultados y aplicaciones son comentados en: